Glossário - Temas de Genética (ATUALIZADO)
PRORROGAÇÃO DO PRAZO:
Neste novo glossário vocês devem adicionar os seus respectivos temas e definições, juntamente com as referências bibliográficas (preferencialmente de livros de Genética).
Prazo: 23/05, ás 18:00h.
A | B | C | D | E | F | G | H | I | J | K | L | M | N | O | P | Q | R | S | T | U | V | W | X | Y | Z | Todos
Critério de ordenação atual: Sobrenome crescente Ordenar por: Sobrenome
DNA extracromossômico | ||||
|---|---|---|---|---|
DNA extracromossômico é o material genético que não faz parte dos cromossomos; unidades de DNA no citoplasma que controlam a herança citoplasmática, como por exemplo o plasmídeo que é transferido de maneira
independente dos cromossomos. (fonte imagem: Só Biologia) | ||||
DNA ligase | ||||
|---|---|---|---|---|
DNA ligase é a enzima que une as extremidades de duas fitas de DNA por meio de uma ligação covalente, formando uma fita de DNA contínua. fonte: ALBERTS, Bruce et al. Molecular biology of the cell. 6th ed. New York: Garland Science, 2015.
(fonte imagem: Science Learning Hub) | ||||
DNA mitocondrial | ||||
|---|---|---|---|---|
O DNA mitocondrial (mtDNA) é o material genético localizado nas mitocôndrias, organelas responsáveis pela produção de energia nas células eucarióticas. Diferente do DNA nuclear, o mtDNA é circular e herdado exclusivamente da mãe. Ele contém genes essenciais para a produção de ATP através da fosforilação oxidativa, além de codificar RNAs e proteínas necessários para a função mitocondrial. fonte: ALBERTS, Bruce et al. Molecular biology of the cell. 6th ed. New York: Garland Science, 2015.  (fonte imagem: Synergy Counselling and Therapy) | ||||
BLBeatriz Laura dos Santos |
|---|
| BL | 37. Cruzamento Teste | |||
|---|---|---|---|---|
CRUZAMENTO- TESTE O cruzamento teste, também conhecido como retrocruzamento, é uma técnica utilizada em genética para determinar o genótipo de um organismo heterozigoto para um gene específico. Esse procedimento envolve o cruzamento do organismo heterozigoto com um homozigoto recessivo para o mesmo gene de interesse. O objetivo principal do cruzamento teste é identificar se o organismo heterozigoto carrega ou não o alelo dominante para o gene em questão. Referências: 1. Griffiths AJF, Miller JH, Suzuki DT, et al. Introdução à Genética. 9ª ed. Guanabara Koogan; 2016. 2. Russell PJ, Hertz PE, McMillan B. Biologia Geral. 6ª ed. McGraw-Hill; 2010. 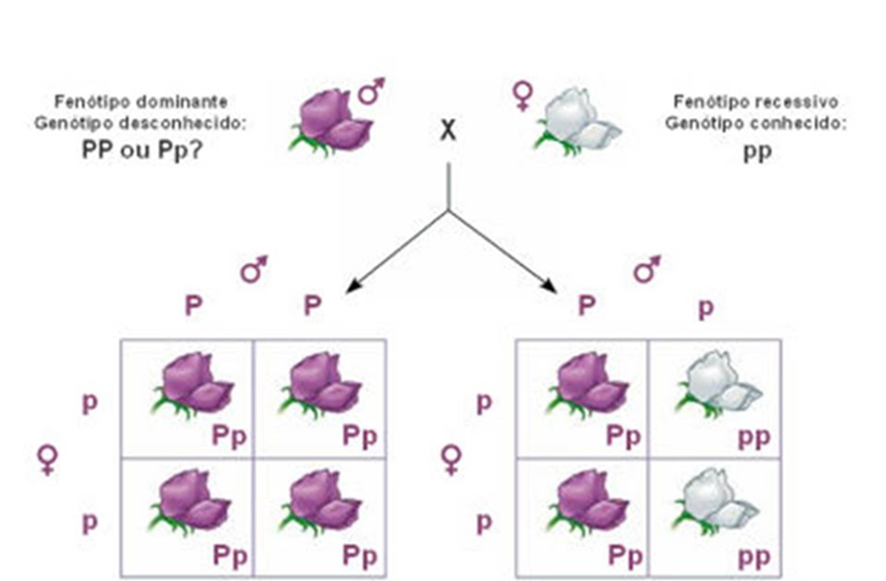 Ref. da Imagem: https://profpenafortehiperlinks.blogspot.com/2012/04/cruzamento-teste.html | ||||
| BL | 38.DELEÇÃO DE BASE | |||
|---|---|---|---|---|
DELEÇÃO DE BASE A deleção de base, também conhecida como deleção de nucleotídeo, é um tipo de mutação genética que envolve a perda de um ou mais pares de bases do material genético. Essa alteração pode ocorrer em diferentes regiões do genoma, incluindo genes codificadores de proteínas, regiões regulatórias e sequências não codificadoras. Características da Deleção de Base:
Exemplo Clínico:
Referências:
 Ref. Imagem: Conselho Regional de Medicina do Paraná https://www.crmpr.org.br/Sindrome-de-Williams-e-tema-de-mostra-fotografica-na-Associacao-Medica-11-50851.shtml | ||||
| BL | 39.DELEÇÃO GENÉTICA | |||
|---|---|---|---|---|
DELEÇÃO GENÉTICA A deleção genética é um termo mais amplo que se refere à perda de uma parte maior do material genético, que pode incluir múltiplos genes, regiões regulatórias e sequências não codificadoras, em contraste com a deleção de base, que envolve a perda de um ou mais pares de bases específicas no DNA. Enquanto a deleção de base é uma forma específica de mutação genética que afeta a sequência de nucleotídeos, a deleção genética abrange alterações mais extensas que podem ter impactos significativos no funcionamento dos genes e no fenótipo do organismo.
Referências:
 Ref. imagem 2: https://vanessacavalcante.com/sindrome-de-prader-willi-p-w-s.html Ref. imagem 2: https://vanessacavalcante.com/sindrome-de-prader-willi-p-w-s.html | ||||
| BL | 40.DESPURINAÇÃO | |||
|---|---|---|---|---|
DESPURINAÇÃO A despurinação é um processo fisiológico que envolve a remoção de purinas do organismo. As purinas são compostos orgânicos nitrogenados encontrados em alimentos e também são produzidas naturalmente pelo corpo durante o metabolismo das células. A despurinação é crucial para manter o equilíbrio adequado de purinas no organismo, evitando assim a acumulação excessiva desses compostos, o que pode levar a condições como gota e cálculos renais. Mecanismos de Despurinação:- Enzimas Despurinadoras: O principal mecanismo de despurinação envolve enzimas específicas que catalisam a conversão de purinas em metabólitos inofensivos que podem ser excretados pelo corpo. Referências: 1. Rodwell VW, Bender DA, Botham KM, Kennelly PJ, Weil PA. Harper's Illustrated Biochemistry. 31st ed. New York: McGraw-Hill Education; 2018. 2. Becker MA, Jolly M. Hyperuricemia and associated diseases. Rheum Dis Clin North Am. 2006;32(2):275-293. 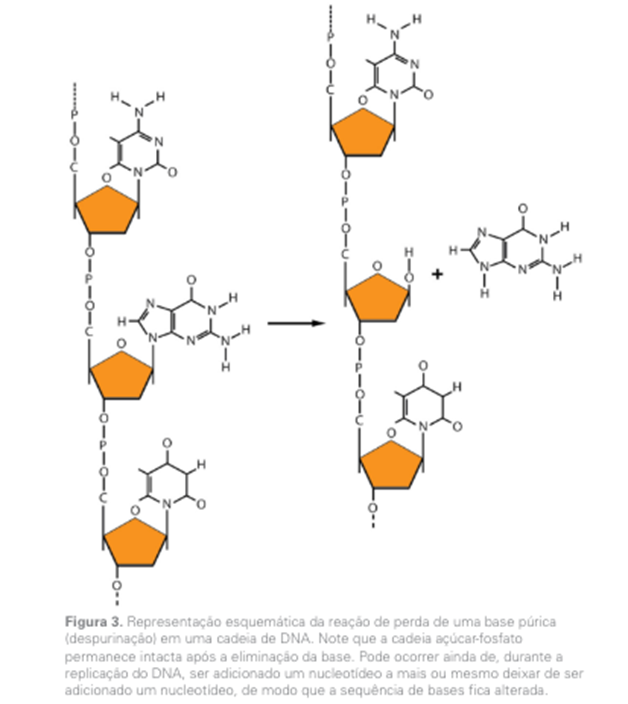 Ref. da Imagem: Biologia Molecular- texto 3-Mutação e Reparo de DNA https://edisciplinas.usp.br/pluginfile.php/3005337/mod_resource/content/1/BiologiaMolecular_texto03%20%283%29.pdf | ||||
Proto-oncogene | ||||
|---|---|---|---|---|
Proto-oncogenes são genes normais que possuem efeitos sobre a proliferação celular. Quando um proto-oncogene sofre uma mutação, ele pode ser ativado, dando origem a um oncogene, que é um gene que faz com que a célula se divida descontroladamente, podendo levar ao surgimento de tumores. Apenas uma única mutação em um alelo pode ser suficiente para ativar um oncogene. Essas mutações que ativam um oncogene podem ocorrer por vários caminhos, podendo ser altamente específicas, causando desregulação ou hiperatividade de uma proteína, e também eventos de amplificação gênica, que geram superabundância do RNAm codificado e do produto proteico. | ||||
Pseudogene | ||||
|---|---|---|---|---|
Pseudogenes são sequências de DNA que se parecem com genes conhecidos, mas não são funcionais e, portanto, não resultam em proteínas funcionais. Os pseudogenes podem ser de dois tipos: processados e não processados. | ||||
Purina | ||||
|---|---|---|---|---|
Purina é uma classe das bases nitrogenadas, sendo um composto orgânico formado por aneis heterocíclicos de carbono e por átomos de nitrogênio. Duas das bases nitrogenadas dos ácidos nucleicos, adenina e guanina, são derivadas de uma purina. | ||||
Quimera genética | ||||
|---|---|---|---|---|
A quimera genética é uma condição genética rara, que faz com que um indivíduo possua mais de um tipo de DNA em seu corpo. Indivíduos com essa condição -as chamadas quimeras humanas- geralmente não apresentam características que indiquem a condição genética, mas há relatos de casos de pessoas com quimerismo que apresentam cor de olhos diferentes ou cor de pele diferente em algumas regiões do corpo. O quimerismo pode ocorrer através de causas naturais, como a ligada ao início do desenvolvimento de gêmeos fraternos: algo impede o desenvolvimento de um dos zigotos e ele é absorvido pelo zigoto que sobreviveu, formando uma quimera; e como o microquimerismo: durante a gestação, a mãe passa a carregar o seu DNA e o DNA do bebê que está gerando. Esse fenômeno também pode acontecer de forma artificial, como por intervenção médica para o tratamento de leucemia e de fibrose cística.
| ||||
GLGiovana Leine dos Santos |
|---|
| GL | Genoma Viral | |||
|---|---|---|---|---|
Os vírus são pequenos parasitas intracelulares, submicroscópicos, que contêm DNA ou RNA como genoma protegido por uma capa de proteína codificada pelo vírus chamada capsídeo. O genoma viral é embalado dentro de um capsídeo proteico simétrico, composto por uma única ou múltiplas proteínas, cada uma delas codificando um único gene viral. Devido a esta estrutura simétrica, os vírus podem codificar todas as informações necessárias para a construção de um grande capsídeo usando um pequeno conjunto de genes. Atualmente, foram descritos mais de 4.000 vírus, classificados em 71 famílias. Embora os vírus possuam genomas pequenos, eles apresentam uma enorme diversidade em comparação com plantas, animais e até bactérias. Com relação ao genoma, os vírus são amplamente divididos em vírus de DNA e vírus de RNA. Os vírus DNA e RNA podem ser de fita simples ou dupla, com um arranjo circular, linear ou segmentado. Os vírus DNA e RNA são diferenciados por suas características, como monopartidos ou multipartidos. Qualquer que seja a composição do genoma viral, todos os vírus são parasitas intracelulares obrigatórios, que podem replicar-se apenas dentro das células hospedeiras apropriadas. Os seus genomas codificados devem ser reconhecidos por uma célula hospedeira específica, que será logo parasitada. Para isso, o código genético empregado pelo vírus deve ser compatível ou reconhecido pela célula hospedeira. Figura 1. Estrutura simples e características de um vírus. Chaitanya KV. Structure and Organization of Virus Genomes. Genome and Genomics. 2019 Nov 18:1–30. doi: 10.1007/978-981-15-0702-1_1. PMCID: PMC7119911. | ||||
| GL | Genômica | |||
|---|---|---|---|---|
Só nas últimas décadas os cientistas progrediram da análise de um único ou de um pequeno número de genes de uma só vez para a investigação de milhares de genes, passando do estudo das unidades de herança à investigação do genoma completo de um organismo. A ciência dos genomas, ou "genômica", inicialmente dedicada à determinação de sequências de DNA (a ordem dos nucleotídeos num determinado fragmento de DNA), expandiu-se rapidamente para um nível mais funcional - estudando os perfis de expressão e os papéis de ambos genes e proteínas (Del Giacco; Cattaneo, 2012). A genômica humana, definida como um campo científico que aspira investigar a estrutura, função e interação de todos os genes em todo o genoma humano, promete melhorar o diagnóstico, o tratamento e a prevenção de doenças (Lazaridis; Petersen, 2005). Del Giacco L, Cattaneo C. Introduction to genomics. Methods Mol Biol. 2012;823:79-88. doi: 10.1007/978-1-60327-216-2_6. PMID: 22081340. Lazaridis KN, Petersen GM. Genomics, genetic epidemiology, and genomic medicine. Clin Gastroenterol Hepatol. 2005 Apr;3(4):320-8. doi: 10.1016/s1542-3565(05)00085-6. PMID: 15822036. | ||||
| GL | Genômica Estrutural | |||
|---|---|---|---|---|
A genômica estrutural visa caracterizar estruturalmente a maioria das sequências de proteínas por meio de uma combinação eficiente de experimento e modelação. Ela precisa determinar estruturas proteicas que tenham pelo menos 30% de identidade com as sequências a serem modeladas, considerando que, abaixo de 30%, haveria grande chance de erros significativos atrapalharem o sequenciamento proteico (Chance MR; et al, 2002). A estrutura terciária obtida com a genômica estrutural de uma sequência proteica permite que a compreensão da biologia seja complementada, levando em consideração que fornece um quadro de referência para informações sobre as sequências proteicas derivadas de dados genômicos, a fim de entender a função bioquímica. Assim, a estrutura permite que o pesquisador atribua uma posição aos resíduos dos aminoácidos, formulando hipóteses sobre como uma determinada proteína funciona. Mas a estrutura oferece mais do que apenas informações sobre proteínas específicas. Também proporciona um meio de comparar proteínas que não apresentam semelhança óbvia ao nível da sequência (Standley DM; et al, 2022). Figura 1. Visão tridimensional de uma estrutura proteica obtida a partir da Genômica Estrutural. Chance MR, Bresnick AR, Burley SK, Jiang JS, Lima CD, Sali A, Almo SC, Bonanno JB, Buglino JA, Boulton S, Chen H, Eswar N, He G, Huang R, Ilyin V, McMahan L, Pieper U, Ray S, Vidal M, Wang LK. Structural genomics: a pipeline for providing structures for the biologist. Protein Sci. 2002 Apr;11(4):723-38. doi: 10.1110/ps.4570102. PMID: 11910018; PMCID: PMC2373525. Standley DM, Nakanishi T, Xu Z, Haruna S, Li S, Nazlica SA, Katoh K. The evolution of structural genomics. Biophys Rev. 2022 Dec 15;14(6):1247-1253. doi: 10.1007/s12551-022-01031-8. PMID: 36536641; PMCID: PMC9753067. | ||||
| GL | Genômica Funcional | |||
|---|---|---|---|---|
A genômica funcional é o estudo de como o genoma e seus produtos, incluindo RNA e proteínas, funcionam e interagem para afetar diferentes processos biológicos. O campo da genômica funcional inclui transcriptômica, proteômica, metabolômica e epigenômica, pois todas elas se relacionam à decodificação do genoma, levando à expressão de fenótipos específicos. Ao estudar genomas inteiros – genômica clínica, transcriptomas e epigenomas – a genômica funcional permite a exploração da relação diversa entre genótipo e fenótipo, não só para os humanos como espécie, mas também para os indivíduos, permitindo uma compreensão e avaliação de como o genoma funcional “contribui” para diferentes doenças.
Carpenter S, Conlan RS. Clinical Functional Genomics. Cancers (Basel). 2021 Sep 15;13(18):4627. doi: 10.3390/cancers13184627. PMID: 34572854; PMCID: PMC8465855. | ||||
GLGisele Lima de Oliveira |
|---|
| GL | GENÓTIPO HAPLÓIDE | |||
|---|---|---|---|---|
GENÓTIPO HAPLÓIDE O genótipo haploide refere-se à composição genética de um organismo que é caracterizado pela presença de apenas um conjunto completo de cromossomos em suas células. Em um organismo haploide, cada célula possui apenas uma cópia de cada cromossomo, resultando em um genótipo que é determinado por essa única cópia de genes. Isso contrasta com organismos diploides, que possuem dois conjuntos completos de cromossomos e, portanto, têm dois alelos para cada gene. O genótipo haploide é fundamental em organismos como fungos e em certas fases do ciclo de vida de plantas e animais, como nas células reprodutivas (gametas). Referência bibliográfica: Thompson & Thompson genética médica. 8. ed. Rio de Janeiro: Elsevier, 2016, 24 e 25. | ||||
| GL | GENÓTIPO MUTANTE | |||
|---|---|---|---|---|
GENÓTIPO MUTANTE O genótipo mutante refere-se à presença de uma alteração ou mutação em um ou mais genes específicos de um organismo em comparação com o genótipo padrão ou selvagem. Uma mutação é uma mudança na sequência de nucleotídeos do DNA, que pode resultar em uma variação na função ou na expressão do gene afetado. O genótipo mutante pode levar a características fenotípicas diferentes das observadas em indivíduos com o genótipo selvagem e pode ter implicações significativas na fisiologia, no desenvolvimento e na adaptação do organismo ao seu ambiente. As mutações genéticas podem ocorrer espontaneamente ou serem induzidas por agentes externos, como radiação ou certos produtos químicos. Estudos sobre genótipos mutantes são importantes para compreender os mecanismos genéticos subjacentes a uma variedade de condições e doenças, bem como para investigar vias metabólicas, processos de desenvolvimento e evolução. Referência bibliográfica: Thompson & Thompson genética médica. 8. ed. Rio de Janeiro: Elsevier, 2016, 24 e 25. | ||||
| GL | GENÓTIPO POLIPLOIDE | |||
|---|---|---|---|---|
GENÓTIPO POLIPLOIDE O genótipo poliploide refere-se a organismos que possuem mais de dois conjuntos completos de cromossomos em suas células. Enquanto organismos diploides têm dois conjuntos completos de cromossomos (2n), os organismos poliploides podem ter três (triploides, 3n), quatro (tetraploides, 4n), ou mais conjuntos de cromossomos. A poliploidia pode ocorrer naturalmente em alguns organismos, mas também pode ser induzida artificialmente em laboratório. Organismos poliploides podem exibir características fenotípicas diferentes de seus parentais diplóides, podendo apresentar vantagens em termos de vigor, resistência a doenças ou adaptação a diferentes ambientes. Referência bibliográfica: Thompson & Thompson genética médica. 8. ed. Rio de Janeiro: Elsevier, 2016, 24 e 25. | ||||
| GL | GENÓTIPO SELVAGEM | |||
|---|---|---|---|---|
GENÓTIPO SELVAGEM O genótipo selvagem refere-se à forma não mutada ou padrão de um organismo em seu ambiente natural. É a composição genética original de uma população ou espécie antes de qualquer manipulação ou seleção artificial. O genótipo selvagem é frequentemente usado como ponto de referência em estudos genéticos e experimentos, especialmente quando se investigam mutações ou variantes genéticas específicas que ocorrem em uma população. Comparar características fenotípicas e genotípicas de organismos mutantes com aqueles do genótipo selvagem pode ajudar a entender o papel de genes específicos em processos biológicos, desenvolvimento e adaptação evolutiva. Referência bibliográfica: Thompson & Thompson genética médica. 8. ed. Rio de Janeiro: Elsevier, 2016, 24 e 25. | ||||
JLJúlia Limberti Gomes |
|---|
| JL | Ligase | |||
|---|---|---|---|---|
É uma enzima envolvida na duplicação do DNA e promove a ligação entre os nucleotídeos de duas moléculas, uma vez que é essencial para essa síntese. DNA ligase é uma enzima de adesão de DNA. Se dois pedaços de DNA tiverem terminações complementares, a ligase pode ligá-las para formar uma molécula de DNA única e contínua. Referência: https://pt.khanacademy.org/science/biology/biotech-dna-technology/dna-cloning-tutorial/a/restriction-enzymes-dna-ligase#:~:text=DNA%20ligase%20%C3%A9%20uma%20enzima,de%20DNA%20%C3%BAnica%20e%20cont%C3%ADnua. | ||||
| JL | Locus | |||
|---|---|---|---|---|
Posição ocupada pelo gene no cromossomo. Formas diferentes de genes (alelos) podem ocupar o locus. Referência: Thompson & Thompson Genética Médica https://g.co/kgs/ZB6paCp | ||||
| JL | Long non coding RNA | |||
|---|---|---|---|---|
RNA não codificante longo (lncRNAs), que são definidos como RNA com mais de 200 nt de comprimento, mas sem capacidade de codificação de proteínas, é cada vez mais enfatizado devido à sua função biológica. No genoma dos mamíferos, mais de 1000 nt de grandes RNAs intergênicos não codificantes (lnc) são claramente conservados em diferentes mamíferos e, portanto, seus padrões funcionais e de expressão gênica implicaram que esses lncRNAs auxiliam em diversos processos biológicos, incluindo regulação do ciclo celular, vigilância imunológica e pluripotência de células-tronco embrionárias. Referência: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC10149988/ | ||||
| JL | Mapeamento genético | |||
|---|---|---|---|---|
É a forma de analisar a sequência do genoma humano de maneira geral. Além disso, é usado para identificar a localização dos genes e suas distâncias. São as posições relativas dos genes ao longo dos cromossomos, como mostrado pela análise de ligação. Referência: Thompson & Thompson Genética Médica https://g.co/kgs/ZB6paCp | ||||
MLMaria Luisa Eid Martins |
|---|
| ML | Recombinação homóloga | |||
|---|---|---|---|---|
A recombinação homóloga acontece entre moléculas de DNA cujos nucleotídeos são semelhantes, sendo comum em cromossomos homólogos e tendo a participação de enzimas recombinatórias. Essa recombinação é capaz de aumentar a diversidade genética e de reparar danos na molécula de DNA, conseguindo estabilizar o genoma. A recombinação homóloga funciona como um reparo de quebra de fitas duplas do DNA, sem perder as informações genéticas contidas nelas, além de permitir a troca de informações na meiose. Através da recombinação homóloga é possível acontecer a conversão gênica, pois a RH pode fazer uma transferência unilateral da informação genética, convertendo os alelos de um cromossomo homólogo na forma de alelo do outro cromossomo. As extremidades 5’ das fitas quebradas são removidas por uma nuclease; através de enzimas, a extremidade 3’ da fita quebrada se insere na fita de DNA homólogo que não está quebrada e procura por uma sequência complementar a ela, que será utilizada como molde. Assim, a fita invasora é alongada por um DNA-polimerase, sendo restaurada, e forma um pareamento de bases que une as duas fitas duplas de DNA. Por fim, é inserido um DNA adicional nas extremidades 3’ das duas fitas, resultado em duas fitas de DNA intactas. Referências Alberts; Bray; Hopkin; Johnson; Lewis; Raff; Roberts; Walter. Fundamentos da Biologia Celular. 4. ed. Porto Alegre: Artmed, 2017. Disponível em: <https://edisciplinas.usp.br/pluginfile.php/7704248/mod_resource/content/1/Fundamentos%20da%20Biologia%20Celular%20-%20Cap.6.pdf>.Acesso em: 9 de maio de 2024. FIGUEIREDO, José Edson Fontes; VASCONCELOS, Maria José Vilaça de. Mutagênese Dirigida por Oligonucleotídeos: Bases Históricas e suas Aplicações em Engenharia Genética. 1. ed. Sete Lagoas, 2017. Disponível em: <https://ainfo.cnptia.embrapa.br/digital/bitstream/item/172364/1/doc-217.pdf>. Acesso em: 9 de maio de 2024. | ||||
| ML | Recombinação não homóloga | |||
|---|---|---|---|---|
A recombinação não homóloga acontece entre regiões do cromossomo que não são homólogas, ou seja, onde não há uma sequência de nucleotídeos semelhantes, sendo comum em cromossomos translocados. Sua função é promover o reparo de danos físicos na molécula de DNA através da junção das duas extremidades da fita de DNA, fazendo-o de modo aleatório. Na recombinação não homóloga é possível haver falhas no reparo do material genético devido à falta de uma fita molde para guiar a restauração da fita de DNA. Essa forma de recombinação pode acarretar também em inversões, duplicações ou deleções no cromossomo e em mutações no DNA. Referências FIGUEIREDO, José Edson Fontes; VASCONCELOS, Maria José Vilaça de. Mutagênese Dirigida por Oligonucleotídeos: Bases Históricas e suas Aplicações em Engenharia Genética. 1. ed. Sete Lagoas, 2017. Disponível em: <https://ainfo.cnptia.embrapa.br/digital/bitstream/item/172364/1/doc-217.pdf>. Acesso em: 9 de maio de 2024. | ||||
| ML | Recorrência | |||
|---|---|---|---|---|
A recorrência indica as chances de um indivíduo ser portador ou não de uma determinada condição genética hereditária. Assim, podem ser realizados diversos testes para verificar tal risco de recorrência, além do aconselhamento genético. É preciso analisar a natureza genética da doença e o heredograma familiar para que seja possível determinar o risco de uma condição se repetir dentro daquela família. Porém, o risco de recorrência é calculado e determinado em uma média, e em cada caso a recorrência pode ser maior ou menor que o estimado. Referências Thompson & Thompson. Genética Médica. 8. ed. Rio de Janeiro: Elsevier, 2016 BERTOLLO, Eny Maria Goloni; CASTRO, Rodrigo; CINTRA, Mariangela Torreglosa Ruiz; PAVARINO, Érika Cristina. O processo de Aconselhamento Genético. Arquivos de Ciências da Saúde, 2013. | ||||
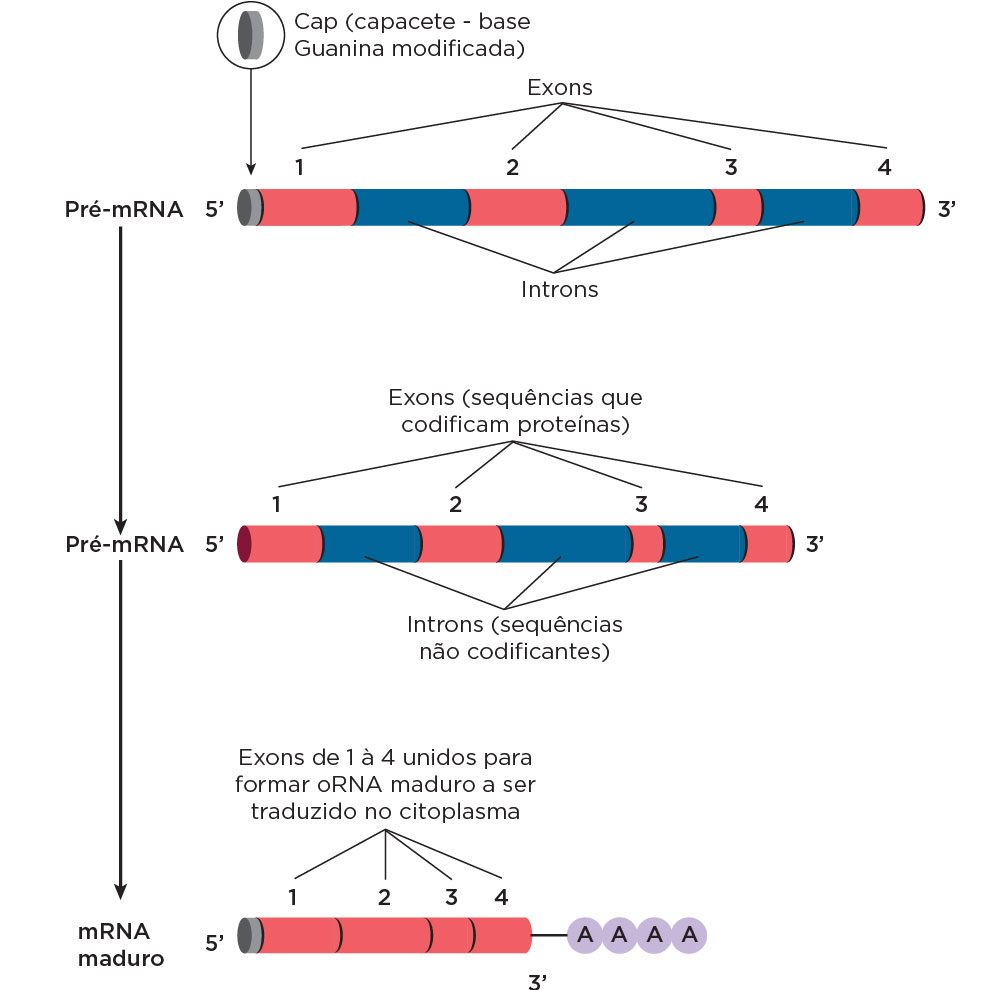
| ML | Região codificadora | |||
|---|---|---|---|---|
A região codificadora de proteínas, também chamada de éxon, forma sequências curtas, com cerca de 120 nucleotídeos. Os éxons são partes importantes do DNA, pois são capazes de codificar as proteínas presentes no RNA mensageiro maduro, englobando, então, as informações essenciais para a síntese do mesmo. Os segmentos dessa região se unem e formam o RNA mensageiro, equivalente à etapa final da transcrição. Dessa forma, os éxons codificam aminoácidos, que se unem e formam uma proteína.
Referências AST, Gil. Genoma Alternativo. Scientifc American Brasil. Disponível em: <https://www2.icb.ufmg.br/grad/genetica/artigo-%20splicing%20alternativo.pdf>. Acesso em: 9 de maio de 2024. NIH - National Human Genome Research Institute. Exon. Disponível em: <https://www.genome.gov/genetics-glossary/Exon>. Acesso em: 9 de maio de 2024. | ||||
ALAna Luisa Rocha da Silva |
|---|
| AL | Bioinformática | |||
|---|---|---|---|---|
É a fusão da biologia com a informática, atua em parceria com a ciência da computação, estatística, matemática e engenharias para analisar, interpretar e processar dados biológicos. Através da bioinformática os cientistas têm desenvolvido novos métodos de mineração de dados dessa grande fonte para aumentar nossa compreensão das células. Em particular, ferramentas de bioinformática têm sido combinadas com tecnologias de robótica, entre outras, para permitir que milhares de proteínas sejam investigadas em um único conjunto de experimentos. Referência bibliográfica: Alberts, B., Johnson, A., Lewis, J., Morgan, D., Raff, M., Roberts, K., & Walter, P. Biologia Molecular da Célula. Artmed Editora. 2017. 6° Edição | ||||
| AL | Capsídeo | |||
|---|---|---|---|---|
Envoltório proteico de um vírus, formado pela auto associação de uma ou mais subunidades proteicas, gerando estruturas geometricamente regulares, que protegem o ácido nucleico viral. Referência bibliográfica: Alberts, B., Johnson, A., Lewis, J., Morgan, D., Raff, M., Roberts, K., & Walter, P. Biologia Molecular da Célula. Artmed Editora. 2017. 6° Edição Fonte imagem: Fig 3-28: Livro: Biologia Molecular da Célula - 6° Edição | ||||
| AL | Cariótipo | |||
|---|---|---|---|---|
Representação do conjunto total de cromossomos de uma célula, organizado de acordo com o tamanho, a forma e o número. A representação dos 46 cromossomos mitóticos é chamada de cariótipo humano. Referência bibliográfica: Alberts, B., Johnson, A., Lewis, J., Morgan, D., Raff, M., Roberts, K., & Walter, P. Biologia Molecular da Célula. Artmed Editora. 2017. 6° Edição Fonte imagem: https://www.biologianet.com/genetica/cariotipo.htm | ||||
| AL | Cauda Poli-A | |||
|---|---|---|---|---|
A cauda poli-A é uma característica encontrada no final (extremidade 3') do RNA mensageiro (mRNA) e é formada por uma série de nucleotídeos adenina (A) adicionados em uma sequência de poli-adenina. Essa cauda desempenha um papel importante na estabilidade e na regulação da expressão gênica. A adição da cauda poli-A ocorre durante o processamento do pré-mRNA após a transcrição do DNA em RNA. Esse processo, chamado poliadenilação, envolve a adição de cerca de 200 a 250 adeninas à extremidade 3' do RNA, facilitada por enzimas específicas. Essa cauda tem a função de atuar como acentuadora da tradução, proteger o mRNA da digestão por nucleases presentes no meio e proporcionar uma maior estabilidade à molécula. Explicação da imagem: A maioria dos mRNAs possui uma sequência de resíduos de adenina na sua extremidade 3’ que é chamada de cauda poli-A (aproximadamente 200 A) e é adicionada à molécula após a transcrição pela enzima poliA polimerase. Quando se reconhece a sequência AAUAAA, altamente conservada localizada 10 a 30 nucleotídeos “upstream” ao sítio de polimerização, é um sinal que a molécula está terminando e que deve ser adicionada a cauda poliA à extremidade da mesma. Referência bibliográfica: Alberts, B., Johnson, A., Lewis, J., Morgan, D., Raff, M., Roberts, K., & Walter, P. (2017). 6° Edição. Biologia Molecular da Célula. Artmed Editora. | ||||