Glossário - Temas de Genética (ATUALIZADO)
PRORROGAÇÃO DO PRAZO:
Neste novo glossário vocês devem adicionar os seus respectivos temas e definições, juntamente com as referências bibliográficas (preferencialmente de livros de Genética).
Prazo: 23/05, ás 18:00h.
Caratteri speciali | A | B | C | D | E | F | G | H | I | J | K | L | M | N | O | P | Q | R | S | T | U | V | W | X | Y | Z | TUTTI
Pagina: 1 2 3 (Successivo)
TUTTI
R |
|---|
| ML | Recombinação homóloga | |||
|---|---|---|---|---|
A recombinação homóloga acontece entre moléculas de DNA cujos nucleotídeos são semelhantes, sendo comum em cromossomos homólogos e tendo a participação de enzimas recombinatórias. Essa recombinação é capaz de aumentar a diversidade genética e de reparar danos na molécula de DNA, conseguindo estabilizar o genoma. A recombinação homóloga funciona como um reparo de quebra de fitas duplas do DNA, sem perder as informações genéticas contidas nelas, além de permitir a troca de informações na meiose. Através da recombinação homóloga é possível acontecer a conversão gênica, pois a RH pode fazer uma transferência unilateral da informação genética, convertendo os alelos de um cromossomo homólogo na forma de alelo do outro cromossomo. As extremidades 5’ das fitas quebradas são removidas por uma nuclease; através de enzimas, a extremidade 3’ da fita quebrada se insere na fita de DNA homólogo que não está quebrada e procura por uma sequência complementar a ela, que será utilizada como molde. Assim, a fita invasora é alongada por um DNA-polimerase, sendo restaurada, e forma um pareamento de bases que une as duas fitas duplas de DNA. Por fim, é inserido um DNA adicional nas extremidades 3’ das duas fitas, resultado em duas fitas de DNA intactas. Referências Alberts; Bray; Hopkin; Johnson; Lewis; Raff; Roberts; Walter. Fundamentos da Biologia Celular. 4. ed. Porto Alegre: Artmed, 2017. Disponível em: <https://edisciplinas.usp.br/pluginfile.php/7704248/mod_resource/content/1/Fundamentos%20da%20Biologia%20Celular%20-%20Cap.6.pdf>.Acesso em: 9 de maio de 2024. FIGUEIREDO, José Edson Fontes; VASCONCELOS, Maria José Vilaça de. Mutagênese Dirigida por Oligonucleotídeos: Bases Históricas e suas Aplicações em Engenharia Genética. 1. ed. Sete Lagoas, 2017. Disponível em: <https://ainfo.cnptia.embrapa.br/digital/bitstream/item/172364/1/doc-217.pdf>. Acesso em: 9 de maio de 2024. | ||||
| ML | Recombinação não homóloga | |||
|---|---|---|---|---|
A recombinação não homóloga acontece entre regiões do cromossomo que não são homólogas, ou seja, onde não há uma sequência de nucleotídeos semelhantes, sendo comum em cromossomos translocados. Sua função é promover o reparo de danos físicos na molécula de DNA através da junção das duas extremidades da fita de DNA, fazendo-o de modo aleatório. Na recombinação não homóloga é possível haver falhas no reparo do material genético devido à falta de uma fita molde para guiar a restauração da fita de DNA. Essa forma de recombinação pode acarretar também em inversões, duplicações ou deleções no cromossomo e em mutações no DNA. Referências FIGUEIREDO, José Edson Fontes; VASCONCELOS, Maria José Vilaça de. Mutagênese Dirigida por Oligonucleotídeos: Bases Históricas e suas Aplicações em Engenharia Genética. 1. ed. Sete Lagoas, 2017. Disponível em: <https://ainfo.cnptia.embrapa.br/digital/bitstream/item/172364/1/doc-217.pdf>. Acesso em: 9 de maio de 2024. | ||||
| ML | Recorrência | |||
|---|---|---|---|---|
A recorrência indica as chances de um indivíduo ser portador ou não de uma determinada condição genética hereditária. Assim, podem ser realizados diversos testes para verificar tal risco de recorrência, além do aconselhamento genético. É preciso analisar a natureza genética da doença e o heredograma familiar para que seja possível determinar o risco de uma condição se repetir dentro daquela família. Porém, o risco de recorrência é calculado e determinado em uma média, e em cada caso a recorrência pode ser maior ou menor que o estimado. Referências Thompson & Thompson. Genética Médica. 8. ed. Rio de Janeiro: Elsevier, 2016 BERTOLLO, Eny Maria Goloni; CASTRO, Rodrigo; CINTRA, Mariangela Torreglosa Ruiz; PAVARINO, Érika Cristina. O processo de Aconselhamento Genético. Arquivos de Ciências da Saúde, 2013. | ||||
| ML | Região codificadora | |||
|---|---|---|---|---|
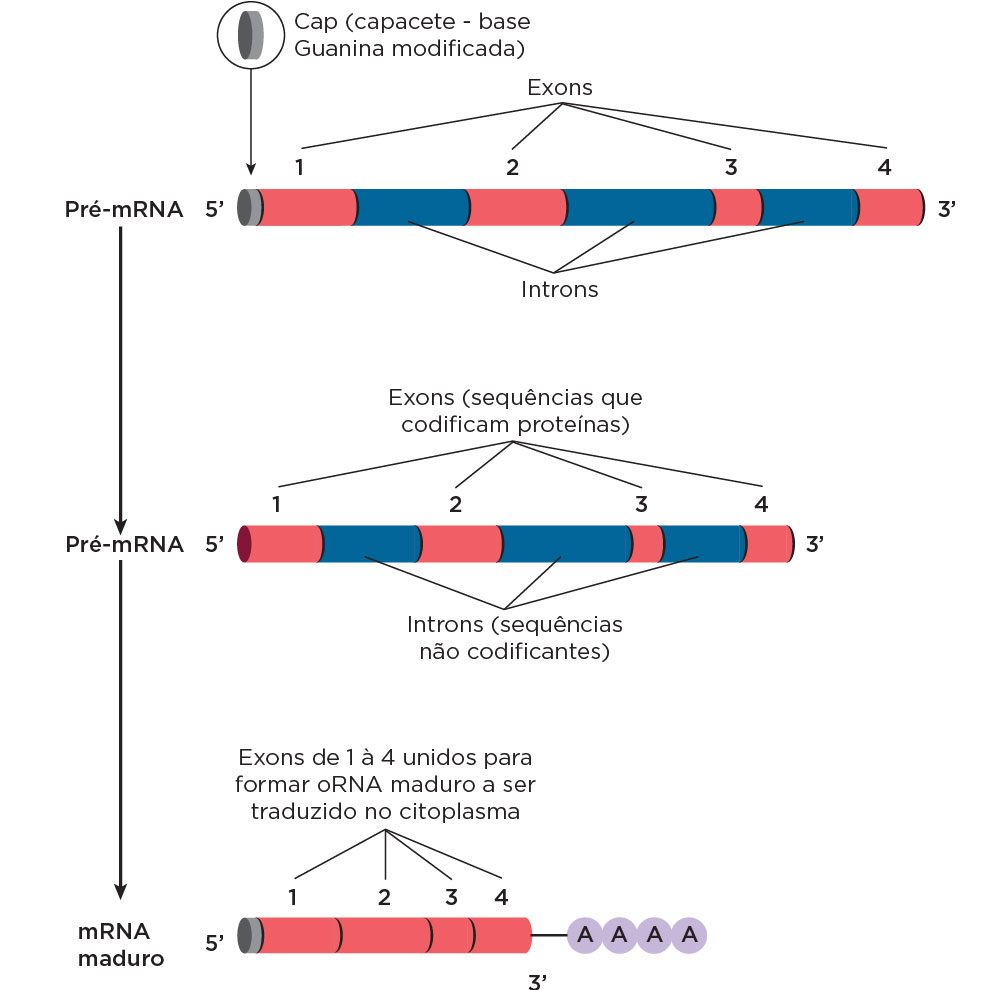
A região codificadora de proteínas, também chamada de éxon, forma sequências curtas, com cerca de 120 nucleotídeos. Os éxons são partes importantes do DNA, pois são capazes de codificar as proteínas presentes no RNA mensageiro maduro, englobando, então, as informações essenciais para a síntese do mesmo. Os segmentos dessa região se unem e formam o RNA mensageiro, equivalente à etapa final da transcrição. Dessa forma, os éxons codificam aminoácidos, que se unem e formam uma proteína.
Referências AST, Gil. Genoma Alternativo. Scientifc American Brasil. Disponível em: <https://www2.icb.ufmg.br/grad/genetica/artigo-%20splicing%20alternativo.pdf>. Acesso em: 9 de maio de 2024. NIH - National Human Genome Research Institute. Exon. Disponível em: <https://www.genome.gov/genetics-glossary/Exon>. Acesso em: 9 de maio de 2024. | ||||
| MC | Região Enhancer | |||
|---|---|---|---|---|
Região Enhancer Os enhancers, também conhecidos como acentuadores, são segmentos de DNA que aumentam a afinidade da maquinaria de transcrição por um promotor específico. Uma Região Enhancer é uma sequência de DNA que pode aumentar a taxa de transcrição de genes específicos. Essas regiões atuam a distâncias consideráveis do promotor do gene e podem estar localizadas tanto a montante quanto a jusante do gene alvo. Eles funcionam através da interação com fatores de transcrição e outras proteínas reguladoras, facilitando a montagem do complexo transcricional necessário para a expressão gênica eficiente. Alguns enhancers são seletivos para células específicas, regulados por proteínas tecido-específicas que se ligam a enhancers específicos, causando uma curvatura no DNA e facilitando o acesso das proteínas da maquinaria de transcrição ao promotor alvo. Essas regiões desempenham um papel crucial no controle da expressão gênica durante o desenvolvimento, em resposta a estímulos ambientais e na diferenciação celular, contribuindo para determinar quais genes são ativados em diferentes tipos de células e em diferentes estágios do desenvolvimento. 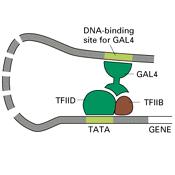 Referência: WATSON, J. D. et al. Molecular Biology of the Gene. 7. ed. Pearson, 2013. | ||||
| MC | Região Silenciadora | |||
|---|---|---|---|---|
Região Silenciadora Uma região silenciadora do DNA refere-se a uma parte específica do genoma onde os genes são suprimidos ou inativados, mesmo que estejam presentes. Essa supressão da expressão gênica ocorre através de modificações epigenéticas, que são alterações químicas na estrutura do DNA ou em proteínas associadas ao DNA, sem alterar a sequência de nucleotídeos. As modificações epigenéticas mais comuns em regiões silenciadoras incluem a metilação do DNA e a modificação das histonas. A metilação do DNA envolve a adição de grupos metil (CH3) em locais específicos do DNA, o que pode interferir na capacidade da maquinaria celular de transcrever genes localizados nesses locais. Por outro lado, a modificação das histonas refere-se à alteração na estrutura das histonas, proteínas ao redor das quais o DNA se enrola no núcleo celular. Diferentes modificações nas histonas podem afetar como o DNA é acessível para a transcrição e, portanto, influenciar a expressão gênica. Além das modificações epigenéticas, fatores como proteínas de ligação ao DNA e RNAs não codificantes também podem contribuir para a formação e manutenção das regiões silenciadoras do DNA. CRAIG, Jeffrey M.; WONG, Nicholas C. Epigenetics: A Reference Manual. Elsevier, 2020. | ||||
| MC | Região Terminadora | |||
|---|---|---|---|---|
Região Terminadora A região terminadora é uma sequência específica de nucleotídeos localizada no DNA que sinaliza o término da transcrição de um gene. Ela desencadeia a dissociação da RNA polimerase, que é a enzima responsável por sintetizar o RNA mensageiro (mRNA) a partir do DNA. A região terminadora geralmente contém elementos como sequências ricas em citosina e guanina seguidas por uma região de estrutura secundária formada por emparelhamento de bases, como um hairpin (grampo) de RNA. Essa estrutura secundária é crucial para o reconhecimento pela RNA polimerase de que chegou ao fim do gene, permitindo que ela se separe do DNA e o mRNA seja liberado. A precisão na terminação da transcrição é essencial para garantir que o mRNA seja completo e funcional na síntese de proteínas durante a tradução. Watson, James D., et al. Biologia Molecular do Gene. 7. ed. Porto Alegre: Artmed, 2014. | ||||
| MC | Relógio Molecular | |||
|---|---|---|---|---|
Relógio Molecular O conceito de relógio molecular refere-se a um sistema intrínseco de cronometragem biológica que permite aos organismos ajustar suas atividades de acordo com o ciclo diário de 24 horas. Este sistema é fundamental para coordenar uma variedade de processos fisiológicos e comportamentais com os padrões ambientais de luz e escuridão. Os relógios moleculares são compostos por um conjunto complexo de proteínas e genes que interagem em ciclos regulares de feedback. Um exemplo comum é encontrado nos ritmos circadianos, que regulam a atividade de genes específicos em resposta aos sinais ambientais, principalmente a luz solar. Esses relógios são autônomos e persistem mesmo em condições isoladas de variações ambientais, indicando que sua operação depende de mecanismos internos. A regulação dos ritmos circadianos envolve processos de transcrição genética, onde certos genes "relógio" são expressos em padrões cíclicos. Esses genes codificam proteínas que se acumulam em níveis específicos ao longo do tempo e, eventualmente, retroagem para controlar sua própria expressão. Esses mecanismos garantem que os organismos possam prever e se adaptar regularmente às mudanças diárias em seu ambiente, otimizando funções biológicas como o metabolismo, a divisão celular, a atividade cerebral e o comportamento. Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., & Walter, P. (2017). Biologia molecular da célula (6a ed.). Artmed. | ||||
| MS | Repetição de Trinucleotídeos | |||
|---|---|---|---|---|
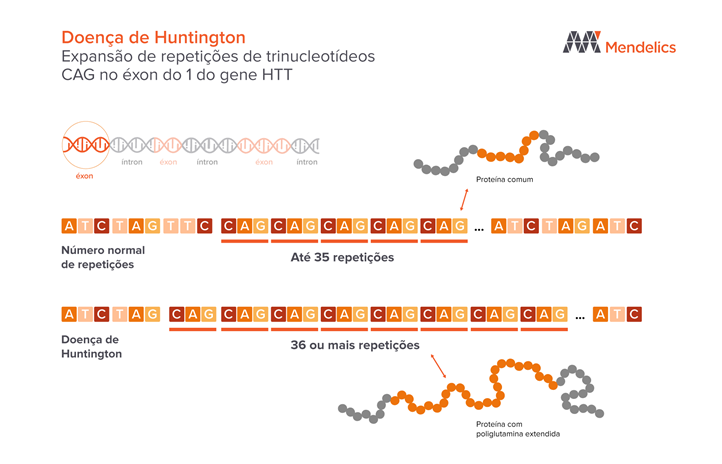
São repetições de três nucleotídeos em tandem (um adjacente ao outro) que podem acontecer por todo o genoma humano e codificar vários aminoácidos. Em geral, os genes associados a essas repetições são selvagens polimórficos, ou seja, já possuem trincas repetidas em baixa quantidade na população normal. O prolongamento dessas repetições é instável (expansões repetidas instáveis) e, ao se expandir, pode levar a doenças (chamadas doenças de Antecipação) neurodegenerativas (mais de 12 confirmadas) como a doença de Huntington, a doença do X Frágil, a Distrofia Miotônica ou a Ataxia de Friedrerich. O aparecimento dessas doenças está relacionado com o fato de que essas repetições de nucleotídeos são hereditárias e podem se acumular a cada geração.
Referência do texto: Genética Médica Thompson Thompson 7Ed | ||||
| MS | Replicação | |||
|---|---|---|---|---|
A Replicação/Duplicação/Autoduplicação é o processo da duplicação da molécula de DNA. Esse processo ocorre durante a interfase (na fase S) do ciclo celular e nele as duas cadeias de nucleotídeos que formam o DNA inicial se separam (pela enzima Helicase que rompe as pontes de hidrogênio) e cada uma delas é pareada (DNA ligase) por uma nova cadeia complementar (DNA polimerase). A replicação do DNA é semiconservativa pois cada molécula de DNA produzida conserva um filamento da molécula de DNA original. Ou seja, a replicação da hélice dupla do DNA, resulta em duas moléculas filhas idênticas, cada uma é composta de uma fita dos pais e uma nova fita sintetizada. 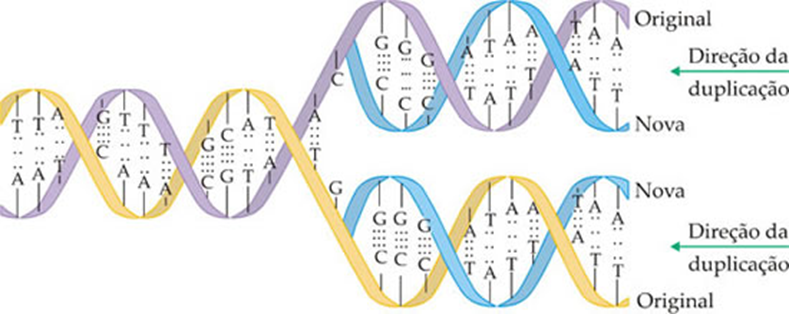 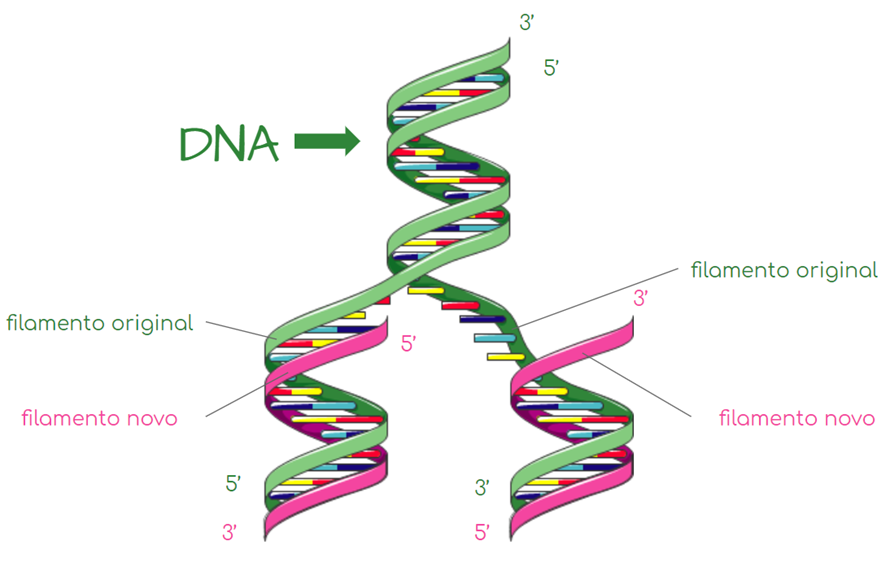 Referência do Texto: Genética Médica Thompson Thompson 7Ed | ||||
Pagina: 1 2 3 (Successivo)
TUTTI