Roteiro da Semana 3
Mapeamento dos cromossomos humanos
Mapeamento dos cromossomos humanos
Os cromossomos de eucariontes contêm grande número de genes, cujas localizações são estabelecidas ao longo de sua extensão. A menos que sejam separados por permutação, os alelos localizados no mesmo cromossomo segregam como uma unidade durante a formação dos gametas. A permutação entre os cromossomos homólogos, durante a meiose, origina gametas recombinantes com diferentes combinações de alelos, que aumentam a variabilidade genética. A permutação entre os cromossomos homólogos funciona como base para a construção de mapas cromossômicos. Quanto maior a distância entre dois genes em um cromossomo, mais alta é a frequência de permutação entre esses genes.
A espécie humana não se presta a estudos de mapeamento cromossômico pelo método utilizado na genética tradicional. Várias dificuldades existem, entre elas: não é possível efetuar cruzamentos de interesse para as pesquisas; o número de descendentes em cada família é, na grande maioria das vezes, muito pequeno; em teoria, o tempo de vida do pesquisador e da espécie em estudo é o mesmo, impedindo a análise de várias gerações.
Os métodos de mapeamento que são ou foram utilizados para a espécie humana incluem: análise de heredogramas, combinação de marcadores gênicos e de marcadores cromossômicos, estudo de aberrações cromossômicas aliadas a fenótipos alterados, hibridação celular e hibridação in situ. Atualmente, esses métodos estão sendo substituídos por técnicas mais moleculares, que surgiram em consequência do advento da tecnologia do DNA recombinante e que serão abordadas com mais detalhes em aulas futuras. Os vários métodos de mapeamento físico oferecem níveis de resolução muito diferentes, desde a localização de um gene em um dado cromossomo como um todo, até a de um gene de sequência única em um ponto específico de um cromossomo.
Análise de heredogramas
A análise de heredogramas permitiu estabelecer ligação entre um número muito pequeno de genes. No entanto, não forneceu nenhuma informação sobre a localização desses genes nos cromossomos. Exceção deve ser feita aos genes localizados no cromossomo X, que eram facilmente identificados devido ao seu padrão de herança.
Polimorfismos cromossômicos
Um marcador cromossômico, que nos idos de 1968 era uma curiosidade citogenética, contribuiu para o mapeamento do gene para o sistema sanguíneo Duffy. Em uma família analisada, um dos alelos desse sistema segregava conjuntamente com um autossomo marcador; era um cromossomo 1 com uma grande constrição secundária na região proximal do braço longo, a qual podia ser identificada em coloração convencional com o corante Giemsa.
Outros marcadores cromossômicos foram úteis para a localização de genes como variantes de bandas C (técnica que identifica regiões de heterocromatina constitutiva), satélites duplos, cromossomos com aberrações estruturais e numéricas. Por exemplo: foi possível relacionar a ocorrência de alterações de tamanho no braço longo do cromossomo 13 e a presença de retinoblastoma; em pacientes com trissomia do cromossomo 21, a atividade da superóxido desmutase solúvel encontra-se aumentada; a primeira indicação para a localização do gene para a distrofia muscular progressiva Duchenne ocorreu através de translocações recíprocas equilibradas envolvendo o cromossomo X e diferentes autossomos.
Hibridação celular
Linhagens celulares de indivíduos com cariótipos ou distúrbios de interesse podem ser congeladas e mantidas em nitrogênio líquido para estudos posteriores.
A partir de 1968, obteve-se um acentuado progresso no mapeamento dos genes humanos e de outras espécies de interesse com o uso da técnica de hibridação de células somáticas; essa técnica implica a fusão em cultura de células de duas espécies distintas, por exemplo, células humanas e células de camundongo ou de chimpanzé. Para a obtenção de células híbridas, é necessário que ocorra a fusão das membranas plasmáticas das células em cultura, o que pode ser conseguido com a utilização de polietilenoglicol ou do vírus Sendai inativado.
Quando as células são de espécies diferentes, geralmente há uma eliminação progressiva dos cromossomos de uma delas. Nas células híbridas humano/camundongo, os cromossomos humanos são preferencialmente perdidos; assim sendo, é possível a obtenção de linhagens com o conjunto cromossômico completo do camundongo e apenas alguns cromossomos humanos. Um determinado gene humano pode ser localizado se o fenótipo por ele determinado estiver presente quando um dado tipo de cromossomo for mantido na célula híbrida, e ausente quando o mesmo cromossomo for perdido.
O primeiro gene localizado por esse método foi o da timidina quinase (TK) no cromossomo 17. Essa enzima é produzida tanto pelas células humanas quanto pelas do camundongo, mas a origem de cada tipo de TK pode ser determinada por eletroforese. Todos os clones analisados que produziam a TK humana retiveram o cromossomo 17. Pelo mesmo método, pode-se localizar, no cromossomo 15, o gene da hexosaminidase A (HEXA), cujas mutações causam a doença de Tay-Sachs de herança autossômica recessiva e com alta frequência nos judeus asquenazim.
Essa técnica de hibridação celular, porém, não permite a localização dos genes em lugares específicos dos cromossomos, nem a determinação da ordem ou da distância entre genes ligados. Essas informações foram obtidas, em alguns casos, a partir da presença de cromossomos com aberrações estruturais.
Hibridação in situ
Hibridações de sondas de DNA ou RNA podem ser executadas diretamente nas células ou tecidos. A hibridação in situ no DNA permite a localização de genes em pontos específicos dos cromossomos. Ela envolve a hibridação de uma sonda de DNA ou de RNA em uma preparação cromossômica em lâmina e visualização do sinal ao microscópio (figura 1).
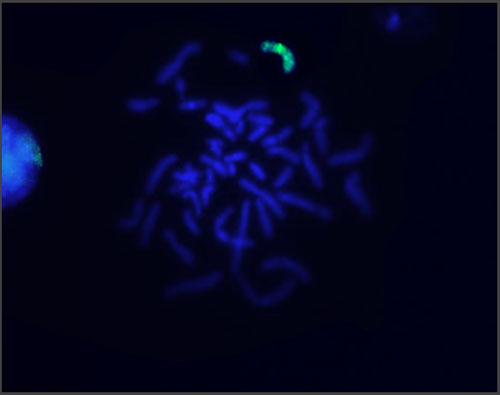
Figura 1 - Hibridação in situ com sondas de DNA.
O DNA presente nos cromossomos é desnaturado na própria lâmina (por isso, a denominação in situ = no local), para que ele possa formar um híbrido com a sonda marcada. A técnica conhecida como FISH (fluorescence in situ hybridization) utiliza sondas marcadas com fluorocromos, compostos que são visualizados em microscópio de fluorescência.
Sondas marcadas por fluorocromos são hibridadas às células fixadas e determinam a localização de uma certa sequência de DNA ou RNA na célula, ou no cromossomo. Enquanto a análise do cariótipo permite visualizar anormalidades da ordem de milhões de pares de bases por microscopia óptica, pela técnica de FISH é possível identificar pequenas alterações cromossômicas, da ordem de dezenas de milhares de nucleotídeos. A FISH pode ser utilizada tanto em células metafásicas como em células interfásicas. Em preparações de cromossomos, uma sonda de interesse para um dado loco autossômico normalmente produzirá dois sinais, cada um deles localizado em uma região específica em um dos cromossomos homólogos. Se uma das cópias da sequência identificada pela sonda contiver uma deleção, o sinal somente será identificado em um dos homólogos. Por outro lado, a presença de mais de 2 sinais sugere uma duplicação do DNA dessa região cromossômica.