Roteiro da Semana 3
| Site: | Moodle USP: e-Disciplinas |
| Cours: | 04 - Genética e Biologia Molecular |
| Livre: | Roteiro da Semana 3 |
| Imprimé par: | Visiteur anonyme |
| Date: | mercredi 26 juin 2024, 12:31 |
Description
Roteiro da semana 3 do curso de Genética e Biologia Molecular.
Introdução
No intervalo de tempo entre os experimentos de Mendel (1865) e sua redescoberta (1900), várias evidências foram acumuladas a partir do interesse que o tema “hereditariedade” despertou no mundo científico. Inúmeras pesquisas foram feitas com o objetivo de localizar os fatores mendelianos, ou seja, o material hereditário na célula. Os avanços em microscopia possibilitaram aos pesquisadores a identificação dos cromossomos e a descoberta de que, na maioria dos eucariontes, os membros de cada espécie têm um número de cromossomos característico (número diploide 2n) na maioria de suas células. Nas células diploides, os cromossomos existem aos pares, sendo que os cromossomos de cada par são denominados homólogos. Os cromossomos homólogos são idênticos quanto ao seu tamanho e localização do centrômero.
Posteriormente, descreveu-se o comportamento dos cromossomos durante duas formas de divisão celular: a mitose e a meiose. As observações das semelhanças entre o comportamento dos cromossomos durante a divisão celular e dos genes durante a formação dos gametas levaram à hipótese de que os genes estão contidos nos cromossomos. Essa proposição foi a base da teoria cromossômica da herança, que explica como as características são transmitidas de geração a geração em uma variedade de organismos, incluindo os humanos.
Teoria cromossômica da herança
Comprovando a teoria
Como os gametas eram as células que faziam a ponte entre uma geração e outra, eles se tornaram o foco das pesquisas. O citoplasma do ovócito tinha um volume muito maior que o do espermatozoide, enquanto os núcleos dessas células tinham tamanhos praticamente iguais; como se acreditava que a contribuição genética de ambos os gametas era semelhante, o núcleo passou a ser o local mais adequado para abrigar o material hereditário.
Em 1902, um graduando de Biologia norte-americano chamado Walter Sutton (1877-1916) e um biólogo alemão, Theodor Boveri (1862-1915), realizaram, independentemente, pesquisas que apontaram os cromossomos como a base física da hereditariedade. Analisando a meiose em uma espécie de gafanhoto, Sutton traçou um paralelo entre a segregação dos homólogos durante a meiose e o comportamento dos fatores mendelianos durante a produção dos gametas em ervilhas. Estudando ovos de ouriço-do-mar, Boveri observou que a ausência de cromossomos alterava o desenvolvimento normal e sugeriu que essas estruturas continham fatores que controlavam o desenvolvimento. Os experimentos realizados em Drosophila melanogaster no laboratório do norte-americano Thomas Hunt Morgan (1866-1945), a partir de 1909, trouxeram a comprovação definitiva da teoria cromossômica da herança.
Genes ligados
Genes ligados
Durante a meiose, ocorre segregação independente dos pares de cromossomos homólogos e, consequentemente, dos genes localizados nesses cromossomos. Entretanto, os genes localizados em um mesmo cromossomo são transmitidos juntos para um mesmo gameta, pois estão ligados, constituindo o que se denomina grupo de ligação. É por causa da ligação gênica (em inglês, linkage) que a lei da segregação independente de Mendel não tem um caráter universal; ela só é válida para genes localizados em diferentes pares de cromossomos homólogos (animação 1 - A e B):
Animação 1 - (A) -> Segregação independente dos genes situados em pares de cromossomos homólogos diferentes; (B) -> Comportamento de genes ligados (A/a e B/b) durante meiose sem permutação.
Morgan e sua equipe analisaram a herança de várias mutações em Drosophila melanogaster e estabeleceram a existência de quatro grupos de ligação. Como essa espécie possui quatro pares de cromossomos homólogos (2n=8 cromossomos), percebe-se uma perfeita correspondência entre esses números, o que constitui uma evidência significativa de que os genes se situam nos cromossomos.
Vamos analisar a seguir um exemplo de ligação gênica estudado por Morgan (animação 2).
Animação 2
Fêmeas selvagens de corpo amarelo e asas normais cruzadas com machos de corpo preto e asas vestigiais originam em F1 apenas descendentes selvagens. Fêmeas selvagens da geração F1, quando cruzadas com machos pretos de asas vestigiais (cruzamento-teste), originam a seguinte descendência:
41,5% amarelos de asas normais
41,5% pretos de asas vestigiais
8,5% amarelos de asas vestigiais
8,5% pretos de asas normais
Como os machos duplo-recessivos produzem um único tipo de gameta pv, esses resultados mostram que as fêmeas F1 duplo-heterozigóticas produzem quatro tipos de gametas em proporções diferentes:
41,5% de gametas PV
41,5% de gametas pv
8,5% de gametas Pv
8,5% de gametas pV
Se os genes considerados segregassem independentemente, como esperado pela segunda lei de Mendel, a proporção obtida seria a mesma para os quatro tipos de gametas (25%). Entretanto, as porcentagens observadas para os gametas PV e pv são bem maiores que as dos gametas Pv e pV.
Os resultados obtidos mostram que os genes para cor de corpo e tamanho de asa estão localizados no mesmo par de cromossomos homólogos, ou seja, estão ligados. Se o alelo P está ligado ao alelo V e o alelo p está ligado ao alelo v, como explicar então na descendência a presença de fenótipos diferentes dos que seriam condicionados por essas combinações de alelos?
Permutação e recombinação
Permutação e recombinação gênica
Os genes que integram um grupo de ligação só podem ser separados através da permutação, fenômeno que ocorre durante a meiose I (animação 3). A permutação é a responsável pela formação de gametas com combinações diferentes dos alelos (gametas recombinantes) que estavam ligados nos cromossomos recebidos dos pais (gametas parentais).
Animação 3
No exemplo anterior, as fêmeas duplo-heterozigóticas formaram dois tipos de gametas em maior porcentagem – que são sempre os parentais – e dois em menor porcentagem – que são sempre os recombinantes. Isso se deve ao fato de que a permutação não ocorre, geralmente, entre os mesmos genes em todas as células que estão se dividindo por meiose. Se os genes estiverem localizados em um mesmo par de homólogos, mas tão distantes que sempre se apresentarão recombinados nos gametas, os resultados vão simular uma segregação independente e a frequência de cada tipo de gameta será de 25%.
Quando há a formação de gametas recombinantes, fala-se em ligação gênica incompleta ou ligação gênica com permutação. A porcentagem de gametas recombinantes permite calcular a taxa de permutação ou taxa de recombinação. No exemplo citado em Drosophila melanogaster, essa taxa seria de 17%, número que corresponde à soma das porcentagens dos gametas recombinantes (8,5% + 8,5%).
Em um indivíduo duplo-heterozigótico, é possível identificar duas disposições distintas dos diferentes alelos de dois genes ligados:
arranjo cis – os alelos dominantes localizam-se em um dos cromossomos e os alelos recessivos, no respectivo cromossomo homólogo;
arranjo trans – o alelo dominante de um gene e o alelo recessivo do outro gene estão presentes no mesmo cromossomo do par de homólogos.
Mapas cromossômicos
Mapas cromossômicos
A recombinação entre genes ligados é o resultado da permutação. Quanto maior a distância entre dois locos gênicos, maior será a taxa de permutação e, consequentemente, maior será a frequência de recombinantes na descendência. Essa proposta, feita pelo grupo de Morgan, pressupunha que os genes apresentavam uma distribuição linear e uma localização constante ao longo dos cromossomos.
Assim sendo, a distância relativa entre dois locos gênicos pode ser calculada a partir da porcentagem de permutação que ocorre entre eles. A taxa de permutação é calculada somando-se as frequências dos indivíduos recombinantes.
Esse é o princípio de construção dos mapas cromossômicos, cuja unidade de medida é a unidade de recombinação (UR) ou centimorgan (cM). No caso dos locos dos genes para cor de corpo e tamanho de asa anteriormente citados, a frequência de recombinação é de 17% e, consequentemente, a distância entre eles é de 17 UR ou 17 cM.
Mapeamento dos cromossomos humanos
Mapeamento dos cromossomos humanos
Os cromossomos de eucariontes contêm grande número de genes, cujas localizações são estabelecidas ao longo de sua extensão. A menos que sejam separados por permutação, os alelos localizados no mesmo cromossomo segregam como uma unidade durante a formação dos gametas. A permutação entre os cromossomos homólogos, durante a meiose, origina gametas recombinantes com diferentes combinações de alelos, que aumentam a variabilidade genética. A permutação entre os cromossomos homólogos funciona como base para a construção de mapas cromossômicos. Quanto maior a distância entre dois genes em um cromossomo, mais alta é a frequência de permutação entre esses genes.
A espécie humana não se presta a estudos de mapeamento cromossômico pelo método utilizado na genética tradicional. Várias dificuldades existem, entre elas: não é possível efetuar cruzamentos de interesse para as pesquisas; o número de descendentes em cada família é, na grande maioria das vezes, muito pequeno; em teoria, o tempo de vida do pesquisador e da espécie em estudo é o mesmo, impedindo a análise de várias gerações.
Os métodos de mapeamento que são ou foram utilizados para a espécie humana incluem: análise de heredogramas, combinação de marcadores gênicos e de marcadores cromossômicos, estudo de aberrações cromossômicas aliadas a fenótipos alterados, hibridação celular e hibridação in situ. Atualmente, esses métodos estão sendo substituídos por técnicas mais moleculares, que surgiram em consequência do advento da tecnologia do DNA recombinante e que serão abordadas com mais detalhes em aulas futuras. Os vários métodos de mapeamento físico oferecem níveis de resolução muito diferentes, desde a localização de um gene em um dado cromossomo como um todo, até a de um gene de sequência única em um ponto específico de um cromossomo.
Análise de heredogramas
A análise de heredogramas permitiu estabelecer ligação entre um número muito pequeno de genes. No entanto, não forneceu nenhuma informação sobre a localização desses genes nos cromossomos. Exceção deve ser feita aos genes localizados no cromossomo X, que eram facilmente identificados devido ao seu padrão de herança.
Polimorfismos cromossômicos
Um marcador cromossômico, que nos idos de 1968 era uma curiosidade citogenética, contribuiu para o mapeamento do gene para o sistema sanguíneo Duffy. Em uma família analisada, um dos alelos desse sistema segregava conjuntamente com um autossomo marcador; era um cromossomo 1 com uma grande constrição secundária na região proximal do braço longo, a qual podia ser identificada em coloração convencional com o corante Giemsa.
Outros marcadores cromossômicos foram úteis para a localização de genes como variantes de bandas C (técnica que identifica regiões de heterocromatina constitutiva), satélites duplos, cromossomos com aberrações estruturais e numéricas. Por exemplo: foi possível relacionar a ocorrência de alterações de tamanho no braço longo do cromossomo 13 e a presença de retinoblastoma; em pacientes com trissomia do cromossomo 21, a atividade da superóxido desmutase solúvel encontra-se aumentada; a primeira indicação para a localização do gene para a distrofia muscular progressiva Duchenne ocorreu através de translocações recíprocas equilibradas envolvendo o cromossomo X e diferentes autossomos.
Hibridação celular
Linhagens celulares de indivíduos com cariótipos ou distúrbios de interesse podem ser congeladas e mantidas em nitrogênio líquido para estudos posteriores.
A partir de 1968, obteve-se um acentuado progresso no mapeamento dos genes humanos e de outras espécies de interesse com o uso da técnica de hibridação de células somáticas; essa técnica implica a fusão em cultura de células de duas espécies distintas, por exemplo, células humanas e células de camundongo ou de chimpanzé. Para a obtenção de células híbridas, é necessário que ocorra a fusão das membranas plasmáticas das células em cultura, o que pode ser conseguido com a utilização de polietilenoglicol ou do vírus Sendai inativado.
Quando as células são de espécies diferentes, geralmente há uma eliminação progressiva dos cromossomos de uma delas. Nas células híbridas humano/camundongo, os cromossomos humanos são preferencialmente perdidos; assim sendo, é possível a obtenção de linhagens com o conjunto cromossômico completo do camundongo e apenas alguns cromossomos humanos. Um determinado gene humano pode ser localizado se o fenótipo por ele determinado estiver presente quando um dado tipo de cromossomo for mantido na célula híbrida, e ausente quando o mesmo cromossomo for perdido.
O primeiro gene localizado por esse método foi o da timidina quinase (TK) no cromossomo 17. Essa enzima é produzida tanto pelas células humanas quanto pelas do camundongo, mas a origem de cada tipo de TK pode ser determinada por eletroforese. Todos os clones analisados que produziam a TK humana retiveram o cromossomo 17. Pelo mesmo método, pode-se localizar, no cromossomo 15, o gene da hexosaminidase A (HEXA), cujas mutações causam a doença de Tay-Sachs de herança autossômica recessiva e com alta frequência nos judeus asquenazim.
Essa técnica de hibridação celular, porém, não permite a localização dos genes em lugares específicos dos cromossomos, nem a determinação da ordem ou da distância entre genes ligados. Essas informações foram obtidas, em alguns casos, a partir da presença de cromossomos com aberrações estruturais.
Hibridação in situ
Hibridações de sondas de DNA ou RNA podem ser executadas diretamente nas células ou tecidos. A hibridação in situ no DNA permite a localização de genes em pontos específicos dos cromossomos. Ela envolve a hibridação de uma sonda de DNA ou de RNA em uma preparação cromossômica em lâmina e visualização do sinal ao microscópio (figura 1).
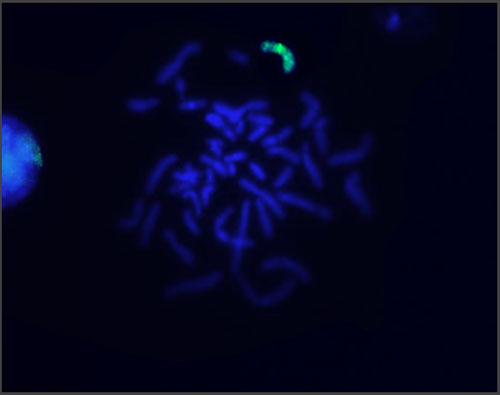
Figura 1 - Hibridação in situ com sondas de DNA.
O DNA presente nos cromossomos é desnaturado na própria lâmina (por isso, a denominação in situ = no local), para que ele possa formar um híbrido com a sonda marcada. A técnica conhecida como FISH (fluorescence in situ hybridization) utiliza sondas marcadas com fluorocromos, compostos que são visualizados em microscópio de fluorescência.
Sondas marcadas por fluorocromos são hibridadas às células fixadas e determinam a localização de uma certa sequência de DNA ou RNA na célula, ou no cromossomo. Enquanto a análise do cariótipo permite visualizar anormalidades da ordem de milhões de pares de bases por microscopia óptica, pela técnica de FISH é possível identificar pequenas alterações cromossômicas, da ordem de dezenas de milhares de nucleotídeos. A FISH pode ser utilizada tanto em células metafásicas como em células interfásicas. Em preparações de cromossomos, uma sonda de interesse para um dado loco autossômico normalmente produzirá dois sinais, cada um deles localizado em uma região específica em um dos cromossomos homólogos. Se uma das cópias da sequência identificada pela sonda contiver uma deleção, o sinal somente será identificado em um dos homólogos. Por outro lado, a presença de mais de 2 sinais sugere uma duplicação do DNA dessa região cromossômica.
[Atividade] - Questionário
![]()
![]() [Questionário] - Para dar sequência às atividades, responda o questionário a seguir.
[Questionário] - Para dar sequência às atividades, responda o questionário a seguir.
